|
|
蛋白质对于人体和生命来说
犹如高楼大厦中的一砖一瓦
失去蛋白质,生命也会如同
缺少材料的大楼般轰然倒塌
人体中存在众多类型的蛋白质,如人体免疫系统的抗体蛋白、胶原蛋白、抗冻蛋白、核糖体等,每种蛋白质都有自己独特的蛋白质三维结构。研究蛋白质,对于生命科学与药物研发具有重要的价值。
很多人体疫病,都是由蛋白质的错误折叠引发的,比如帕金森症、阿尔斯海默症、亨廷顿症等。药物原理类似于一把钥匙,锁就是疫病靶点,通常可以把它认为是一种蛋白质。“钥匙小分子”加入锁孔就是和蛋白质发生结合,抑制蛋白质正常作用,或者激活蛋白质的某些作用。
因此,了解和预测蛋白质的形状,有利于科学家设计出新的更有效的治疗疾病的方法,帮助新药物发现,降低实验成本。
蛋白质是由氨基酸序列组成,但真正决定蛋白质作用的是它的3D结构,即氨基酸序列的折叠方式,如下图的示例。蛋白质结构发现的主要方法,包括X-ray晶体衍射法、核磁共振法,以及2013年后成为热门的冷冻电镜三维重构法等。但是冷冻电镜采购成本高昂,图像重构需要耗费大量的计算力,往往需要很长时间才能解出一个新的蛋白质3D结构。
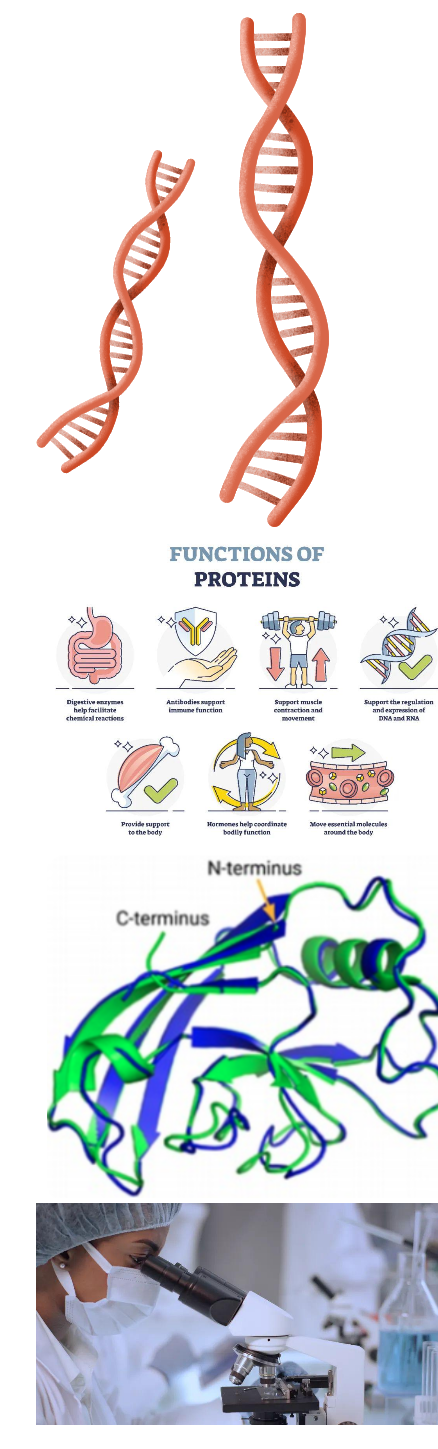
1972年诺贝尔化学奖得主,美国生物化学家克里斯蒂安. 安芬森提出:给定一个氨基酸序列,理论上就能预测出蛋白质的3D结构。五十年来,为了验证这个理论,科学家尝试各种模型预测蛋白质结构的方法,但是在全球蛋白质结构预测领域最知名的CASP竞赛中,直到2018年预测准确率的成绩只是40%上下。
蛋白质越大,模型越复杂和困难,因为需要考虑氨基酸之间更多相互作用。据统计,枚举一个蛋白质可能的构型平均有10的300次幂的搜索空间。采用传统的如分子动力学结构预测计算方案,需要极高的算力以及漫长的计算时间。在过去50年的时间内,只有17%的人类蛋白质组得到结构解析。
2020 年Google DeepMind推出的AlphaFold2改变了一切。2020年12月发布的CASP14成绩单,AlphaFold2将CASP蛋白质结构预测成绩 提高到92.4分(满分100分),与蛋白质真实结构只差一个原子的宽度。2021年7月,Alphafold2模型结构及训练过程发布在Nature杂志,并开源了蛋白质结构数据库及推理代码。
Alphafold2能够预测出98.5%的人类蛋白质结构,其中60%的结构位置预测具有可信度。Science杂志则把AlphaFold2评选为2021年十大科学发现之首。
Alphafold2相较第一代AlphaFold的卷积神经网络,利用多序列比对(MSA),将蛋白质的结构和生物信息整合到深度学习算法,主要包括神经网络EvoFormer和结构模块Structure Module:
EvoFormer主要将图网络和多序列比对结合完成结构预测,图网络将蛋白质相关信息构建成一个图表,以此表示不同氨基酸质间的距离;通过三重Attention自注意力机制来处理氨基酸之间的关系图。结构模块主要将EvoFormer得到的信息转换为蛋白质的3D结构。AlphaFold2是一个端到端神经网络,反复将最终损失应用于输出结果,然后对输出结果进行递归,以不断逼近正确结果。
那么,训练AlphaFold2以及使用AlphaFold2进行蛋白质结构预测的推理计算,需要怎样的计算力支持?戴 尔科技中国研究院以及戴尔数据中心业务部解决方案团队,通过在GitHub下载AlphaFold2模型代码,部署在Dell PowerEdge XE8545服务器上,使用NVIDIA A100 GPU测试AlphaFlod2对68-2750个氨基酸残基组成的不同大小的蛋白质进行3D结构预测,对AlphaFold2的计算性能和特性进行评 估。
戴尔PowerEdge XE8545是戴尔科技最新推出的15G服务器家族中,专门针对AI GPU计算进行设计和优化的加速服务器。4U空间内可以支持4张A100 GPU加速卡,GPU之间通过NVLink实现600GB/s的pear-to-pear高速直连通信。
,时长03:40
测试环境硬件及软件配置如下:
·AMD EPYC 7713 64-Core Processor × 2
·1024 GB memory
·Nvidia A100 GPUs × 4, 80GB/500W
·CentOS Linux 7.0
·Python 3.8.0, TensorFlow 2.5.0
·CUDA 11.5, cuDNN 8.3
XE8545推理68-2750个氨基酸残基组成的蛋白质的3D结构预测耗费的计算时间如下表所示(Top1模型,即推荐置信度最佳的模型),使用单张A100推理计算时间从19.3分钟到2个半小时不等。

通 过性能日志分析,我们可以明显地看到AlphaFold2在推理过程中,由CPU和GPU交替计算,第一阶段同源序列搜索、模版搜索及特征构造,以及最后 阶段3D结构生成的计算过程主要由CPU计算;中间第二个阶段Evoformer神经网络和结构模块计算则主要由GPU进行计算。而XE8545所提供的强劲GPU算力与AMD 多核CPU算力(128核),则能够确保AlphaFold2在规定时间内完成一个大型的蛋白质3D结构的预测计算。
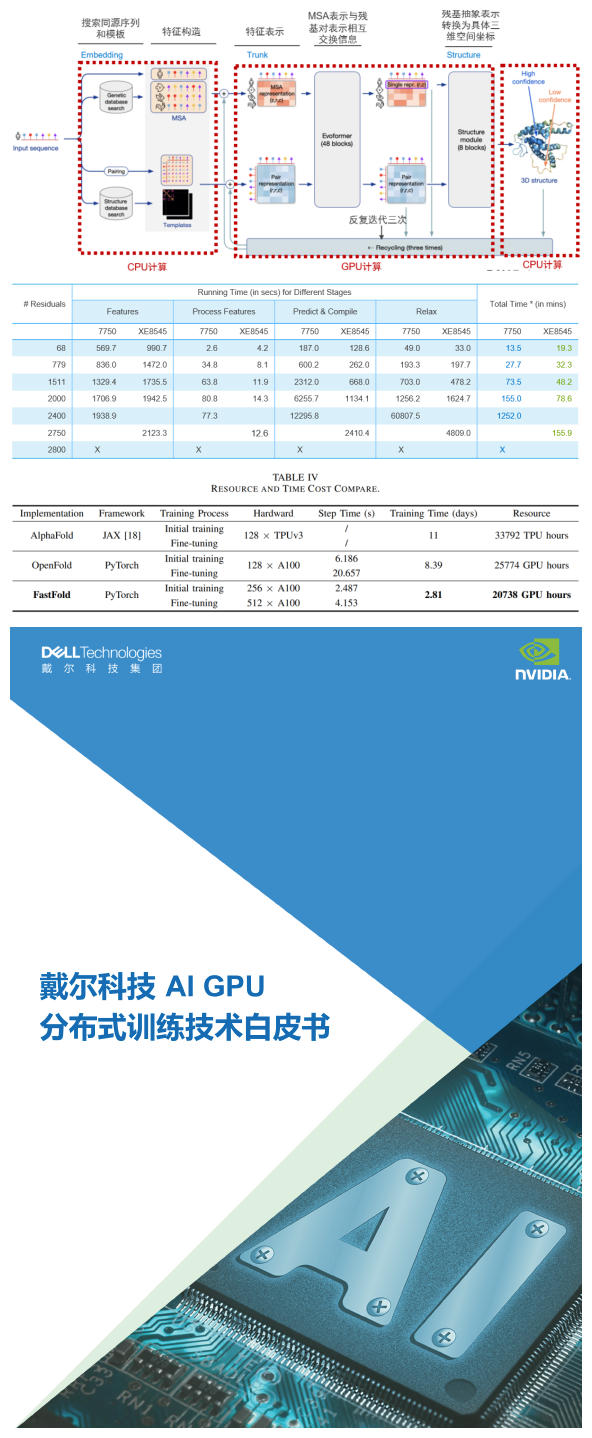
我们也对比了不同GPU对于AlphaFold2推理计算性能的影响。我们选取了一台戴尔7750工作站,配置一张NVIDIA RTX5000显卡,对蛋白质结构预测(Top1模型)计算性能进行对比,对比结果如下表所示:
实验数据显示:当蛋白质规模很小的时候,企业级与消费级GPU性能相差不大;越大的蛋白质,使用A100结构预测加速性能越明显。预测1511个残基的蛋白质3D结构,XE8545+A100耗费时间是RTX5000的65%;预测2000个残基的蛋白质3D结构,XE8545耗费的时间只有RTX5000的50%。
我们可以看到,当预测2800个残基的蛋白质结构时,RTX5000由于显存容量和算力的限制,无法完成结构预测工作,而XE8545仍然以小时级的时间顺利完成同等规模的蛋白质结构预测。
从模型训练的角度来看,Alphafold2以及后续出现的类似的蛋白质结构预测模型,由于采用Transformer机制,模型训练需要非常高的计算力,通常需要64-512张GPU组成计算集群,采用分布式训练机制,才能在比较短的时间内实现模型收敛。
DeepMind 在论文中谈到,训练AlphaFold2模型使用128块Google TPU芯片,接近2周时间完成模型训练。2022年3月,上海交通大学与潞晨科技发布的FastFold模型,使用256张A100 GPU进行初始训练和512张A100进行Fine-tuning,2.81天完成模型训练。
戴尔科技AI GPU分布式训练解决方案,能够提供高速GPU计算、小文件IO快速读写(蛋白质数据库存在大量小文件)和高带宽低延迟地网络通信,帮助用户实现在深度学习框架下分布式训练的自动化实现与性能优化,轻松应对AI时代浪潮。
除此以外,2021年发布的《戴尔科技AI GPU分布式训练技术白皮书》,还可以为用户AI大模型GPU分布式训练提供基础架构解决方案、参考架构及优化建议。公众号后台回复关键字“白皮书”即可轻松获取哦~
|
|
 /2
/2 